목차
<논문링크>
U-Net은 Biomedical 분야에서 이미지 분할(Image Segmentation)을 목적으로 제안된 End-to-End 방식의 Fully-Convolutional Network 기반 모델이다.
22.09 기준 논문 인용 47,000회.
0. Abstract
U-Net은 의료 계열에서의 두 가지 문제 상황을 해결하기 위해서 나왔다.
1.
데이터의 부족
딥러닝은 많은 학습 데이터를 필요로 하지만, biomedical의 특성상 data의 수가 많이 부족할 수 밖에 없다. 그 이유는 환자의 개인정보등에 의한 민감한 문제들로 인하여 Training data를 구하기가 어렵고, 구한다 하더라도 레이블링을 하기 위해서는 일반인이 아닌 전문가들이 직접 해야하기 때문이다.
2.
인접 클래스의 셀 구분
같은 클래스가 인접한 셀 구분이 어려운데 단순 세그멘테이션은 이를 수행하기가 어렵다. 이를 구분하기 위해 인스턴스(셀)의 경계를 테두리로 만들어야 하는 작업이 필요하다.
U-Net은 전반적인 컨텍스트 정보를 얻기 위한 네트워크와 정확한 Localization을 위한 네트워크가 대칭 형태로 구성된다.
본 논문에서는 레이블 정보가 있는(annotated) 데이터가 적을 때 효율적인 데이터 증진(data augmentation) 기법을 이용했다는 점을 강조한다.
1. Introduction
Biomedical image processing 분야의 경우 각각의 픽셀이 클래스를 가지는 "localization"이 필요하다. 또, 앞서 말한 데이터 부족 문제로 인해 라벨된 이미지의 수가 부족하다.
이러한 한계를 극복하기 위해 U-Net 이전에 Sliding Window 방식을 도입하였다.
Sliding Window는 다음과 같은 장점이 존재한다.
•
Localize를 가능하게 한다.
•
패치(Patch) 단위의 학습을 할 경우 데이터의 양이 많아지는 효과가 있다.
•
EM segmentation challenge에서 좋은 결과를 가져온다.
단, 이 경우 픽셀을 모두 예측하는게 아니라 단 중앙 하나의 픽셀에 대해서만 Classification
을 수행한다. 따라서 다음과 같은 단점도 존재한다.
•
각각의 패치에 대해 학습을 하기에 속도가 느리고 패치마다 많은 영역이 겹치는 부분이 생긴다.
•
Localization 정확도와 context의 사용간에 trade-off가 존재한다.
◦
패치가 크면 주변정보도 같이 학습이 가능하지만 이미지의 크기를 줄이기위해 많은 Pooling이 필요하고 이는 localization accuracy을 떨어트린다.
◦
Patch가 작으면 Sub sampling에 의한 정보 손실은 작아지지만 작은 context만 보는 문제점이 있다.
이를 해결해주기 위해, 논문에서는 Sliding Window 방식에서 1개의 픽셀만 Classification하는게 아닌 패치 영역을 Classification하는 특징을 보인다.
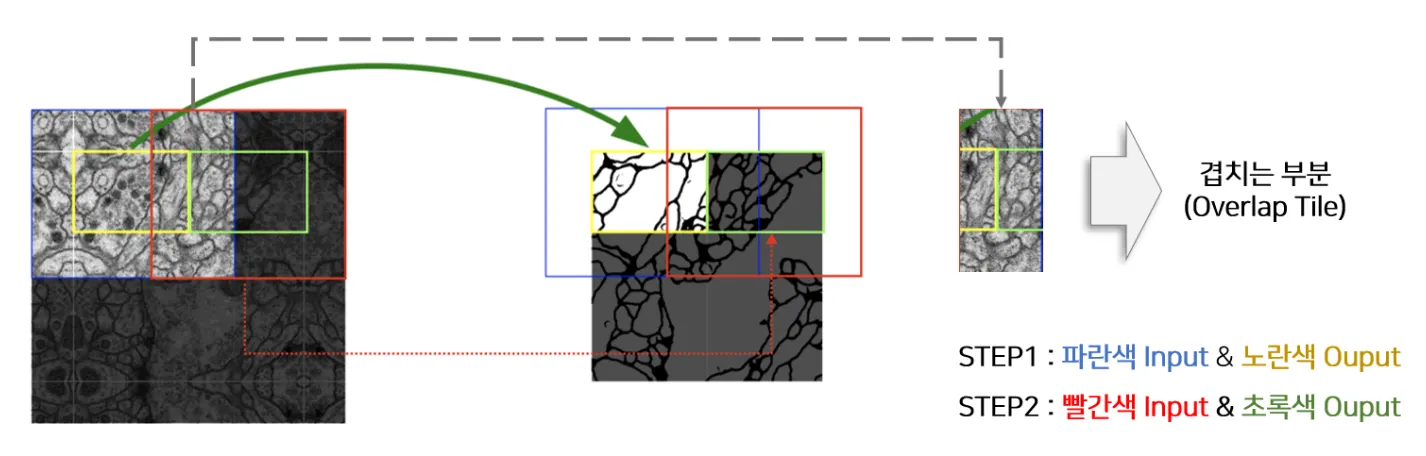
그리고 이때, 전체 영역을 다 예측하는게 아닌 padding을 0으로 줘서 572의 입력을 받아 388 크기의 아웃풋을 생성한다. 이를 Overlap-tile 전략이라 한다.
Overlap-tile
•
FCN(Fully Convolutional Network) 특성상 Fully Connected layers를 사용하지 않고 오직 Convolution만 사용하여 입력 이미지의 해상도에는 제한이 없다. FC layer의 한계점 참고.
•
하지만, U-Net 구조상 출력 이미지의 해상도가 입력 이미지보다 작고, 따라서 Overlab-tile을 사용한다.
•
큰 이미지를 한정된 GPU에서 학습하도록 한다.
다음의 STEP1, STEP2 과정을 진행 후, 파란색과 빨간색 Input이 겹치는 부분을 Overlap Tile이라고 한다. 이때 U-Net의 경우 572의 입력을 388까지 줄이기에 외각 부분의 예측을 수행 시 손실되는 부분이 양쪽으로 92칸의 픽셀만큼 생긴다.
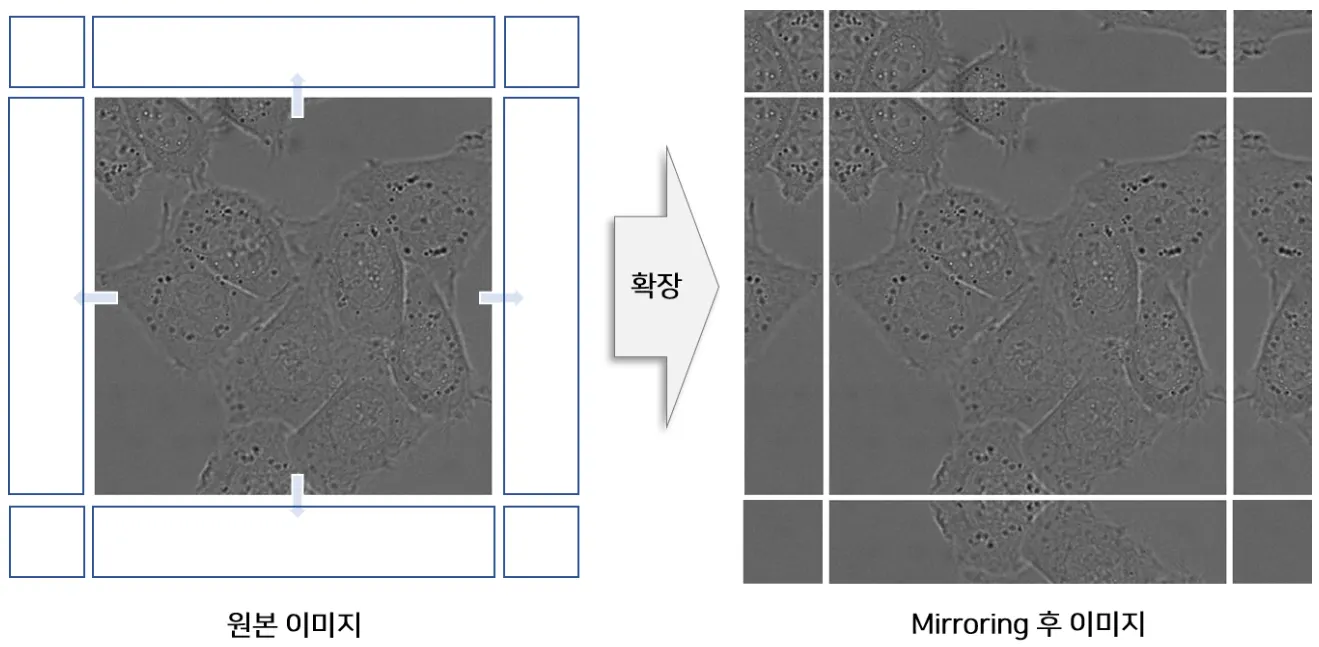
이를 해결하기 위해 논문에서는 외각 부분에 대해 Mirror padding을 적용한다.
Mirror padding은 이미지의 테두리 영역에 픽셀을 예측하기 위해서 입력 이미지를 미러링한 부분을 패딩하는 것이다.
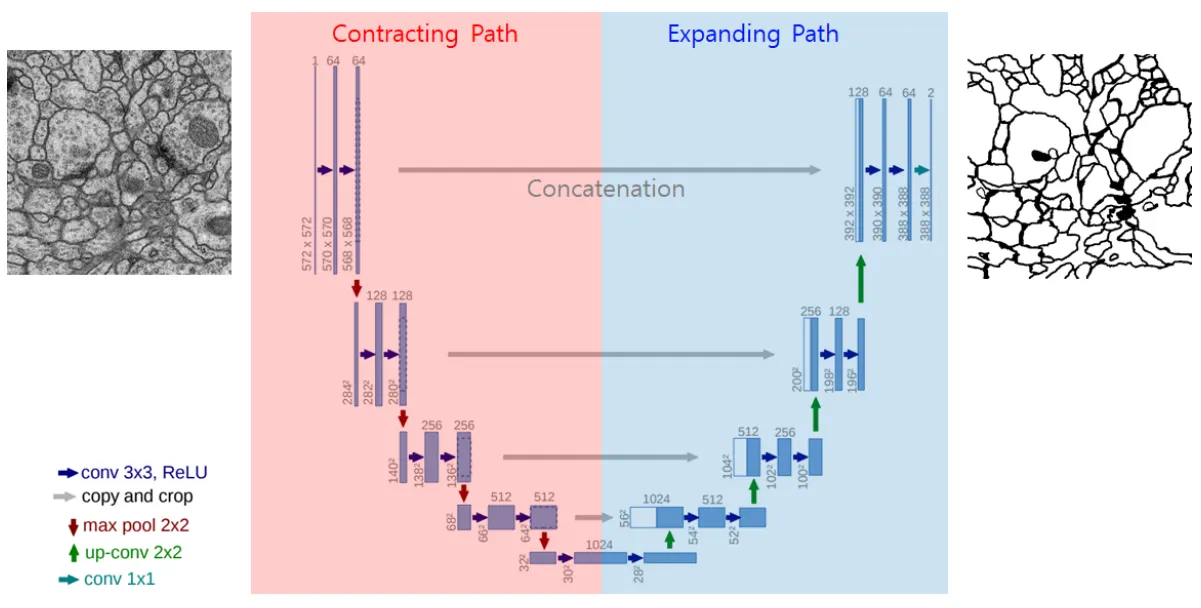
2. Network Architecture
Unet은 FCN을 확장하여 다음과 같은 U자형의 네트워크를 제안했다.
이는 Contracting Path와 Expanding Path를 대칭시킨 것이다.
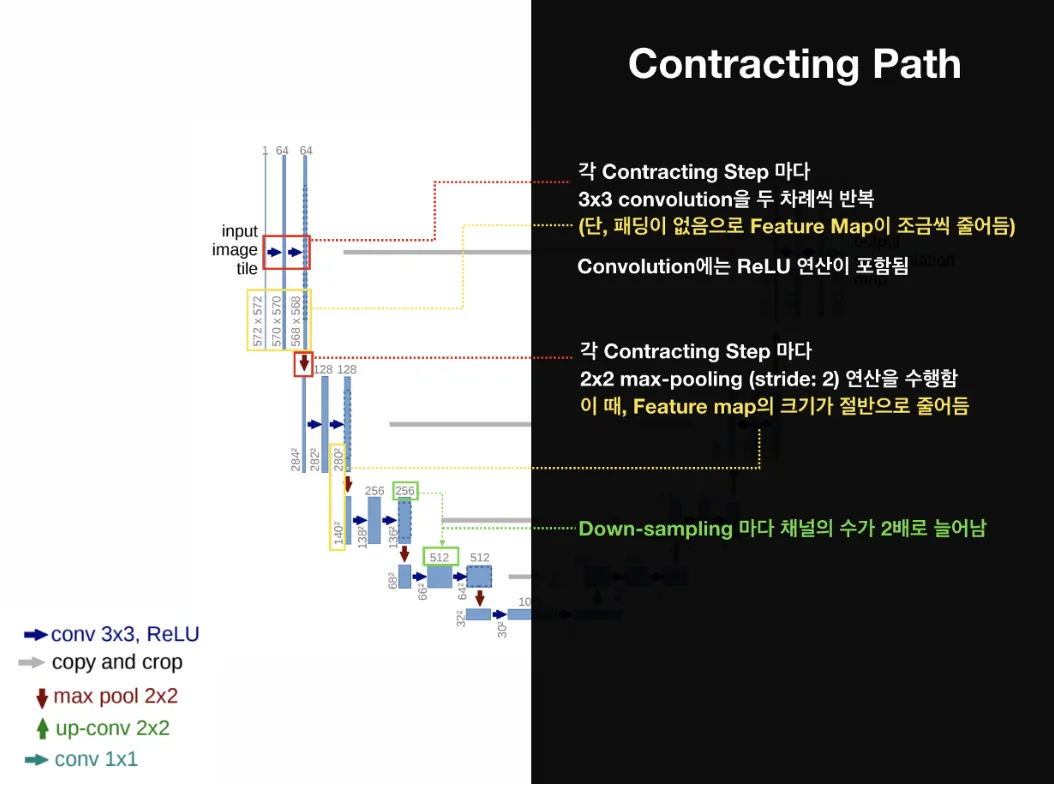
Contracting Path
수축 경로(Contracting Path)는 이미지에 존재하는 넓은 문맥(context) 정보를 처리한다.
이는 일반적인 분류모델에서 사용되는 Down-sampling을 수행하며, Strided Convolution을 사용하여 Feature Map의 너비와 높이를 감소시킨다. 따라서 해상도가 감소한다.
•
U-Net 아키텍처의 초반 부분
•
3x3 Convolution Layer (+ ReLU + BN)을 두 차례씩 반복 (p = 0, s = 1)
◦
Feature Map 해상도 2씩 감소
•
2x2 Max pooling Layer (s = 2)
◦
Feature Map 해상도(너비와 높이) 2배 감소
•
Conv 연산으로 채널 크기는 2배 증가
Bottel Neck
•
3x3 Convolution Layer (+ ReLU + BN)을 두 차례 반복 (p = 0, s = 1)
•
Dropout Layer : 모델을 일반화하고 노이즈에 견고하게(Robust) 만드는 장치
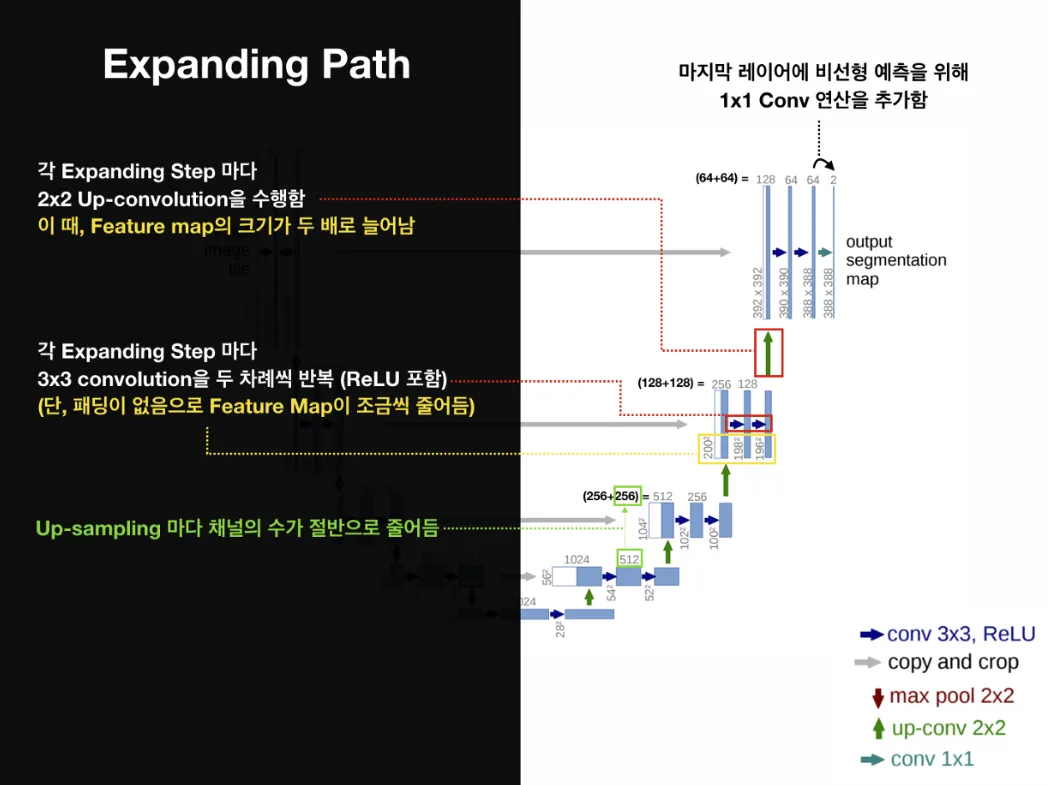
Expanding Path
확장 경로(Expanding Path)는 정밀한 지역화(Precise Localization)가 가능하도록 한다.
이는 Up-sampling을 수행하며, Transposed Convolution을 사용하여 Feature Map의 너비와 높이를 증가시키고 원본 크기로 되돌린다. 따라서 해상도가 증가한다.
•
U-Net 아키텍처의 후반 부분
•
2x2 Deconvolution Layer (s = 2)
◦
Feature Map 해상도(너비와 높이) 2배 증가
•
3x3 Convolution Layer (+ ReLU + BN)을 두 차례씩 반복 (p = 0, s = 1)
•
Conv 연산으로 채널 크기는 2배 감소
•
수축 경로에서 처리된 feature채널 레벨에서 연결(concatenation) map을 잘라내어(cropping) 가져와 수행
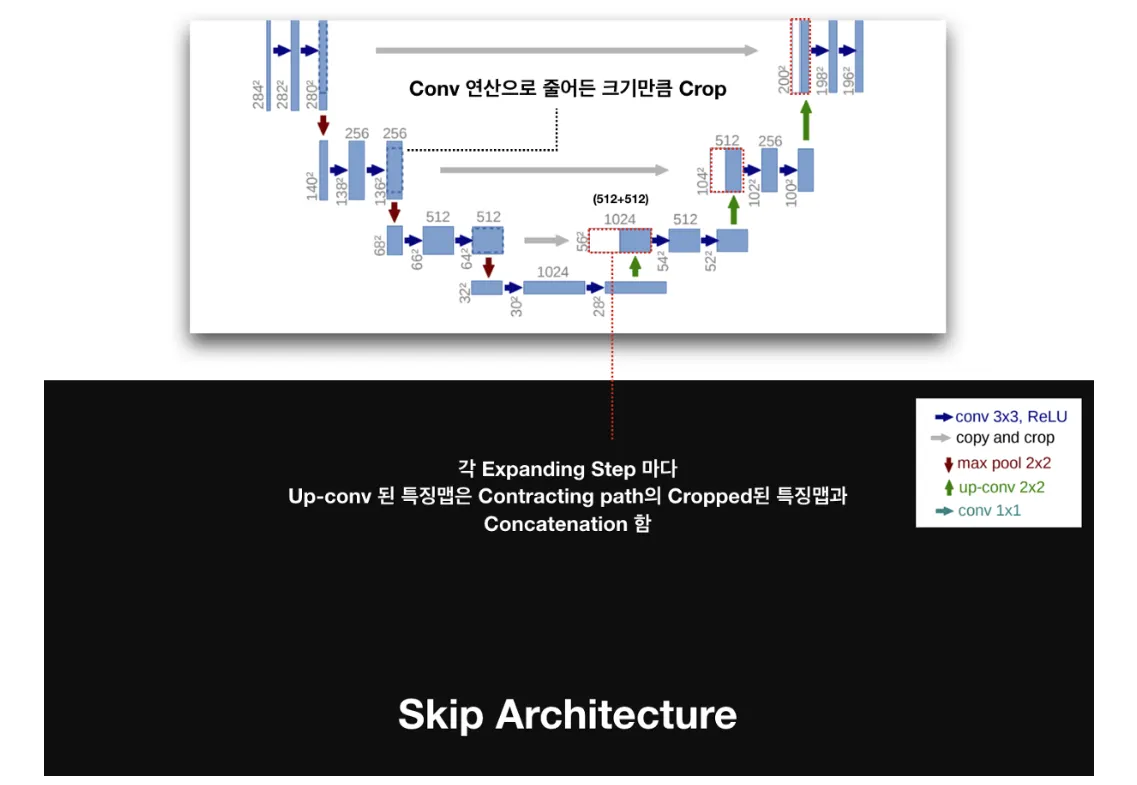
Skip Connection
확장 경로는 Skip Connection을 통해 수축 경로에서 생성된 Feature Map을 잘라내어(cropping) 이를 연결(concatenation)한다. 이를 통해 Contextual 정보와 위치 정보를 결합하는 역할을 한다.
3. Training
•
Optimizer
◦
Stochastic Gradient Descent (SGD)
◦
Momentum : 0.99
•
Deep Learning Framework
◦
Caffe
•
Batch
◦
A single image
3-1. U-Net의 학습 방법 : Objective Function
SoftMax
•
U-Net은 segmentation을 위한 네트워크이므로, 픽셀 단위(pixel-wise)로 소프트맥스(softmax)를 사용한다. 다음은 softmax 공식이다.
: 픽셀의 위치(pixel position)이며,
: 번째 특징 채널(feature channel) = 클래스
: 번째 채널의 위치의 activation 값
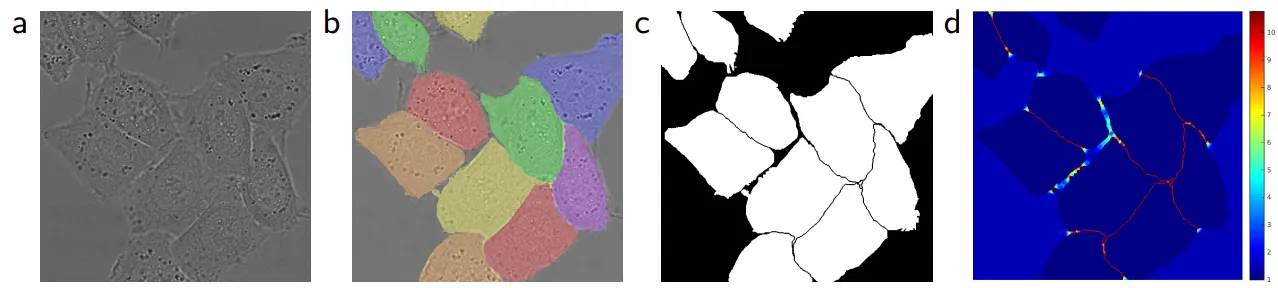
a : 원본이미지 b : 분할 정답 이미지 c : 생성된 분할 마스크 d : 손실함수의 가중치 히트맵
(b)의 다른 색은 서로 다른 인스턴스(instance)를 의미하고, (d)는 경계 픽셀을 학습하기 위해서 픽셀 단위로 강조된 손실함수의 가중치를 의미한다.
Weight Map
•
세포(cell)를 명확히 구분하기 위해 경계 사이에 반드시 배경이 존재하도록 처리하여 작은 분리 경계(small separation border)를 학습한다.
◦
w(x)는 인접한 셀(touching cell) 사이에 있는 배경 레이블에 대하여 높은 가중치를 부여한다. (명확한 분리)
→ 클래스별 빈도에 따른 가중치
→ 가장 가까운 셀과의 거리
→ 두 번째로 가까운 셀과의 거리
위 함수는 과 의 거리의 합이 최대가 되도록 하는 손실함수이다. 이렇게 하면 모델은 낮은 손실함수를 갖는 방향으로 학습하기 때문에 두 세포 사이의 간격을 넓히는 식, 즉 두 인스턴스 사이의 배경을 넓히는 방향으로 학습하게 된다.
Cross-Entropy
•
학습을 위해 다음의 cross-entropy 손실을 사용한다. (true label만 고려하므로, 일반 cross entropy 공식과 동일)
: 이미지의 의 true label
: Featuremap에 있는 각 픽셀
: 추가적인 가중치(weight) 함수 사용
•
Weight Initialize
◦
Gaussian Distribution(가우시안 분포)
▪
Standard deviation :
▪
N = Conv filter size x Conv filter channel
→ 3x3 convolution & 64 feature channel ⇒ N = 3x3x64 = 576
3-2. U-Net의 데이터 증진 (Data Augmentation)
의료 데이터에서는 학습 데이터의 수가 적은 경우가 많으므로, 데이터 증진(data augmentation)이 필요하다.
본 논문에서는 Random Elastic Deformations를 사용했고, 이는 다음과 같은 역할을 한다.
•
모델이 invarinace와 robustness를 학습할 수 있도록 한다.
•
shift and rotaion invariance와 deformations에 대한 robustness, gray value variations을 충족시킨다.
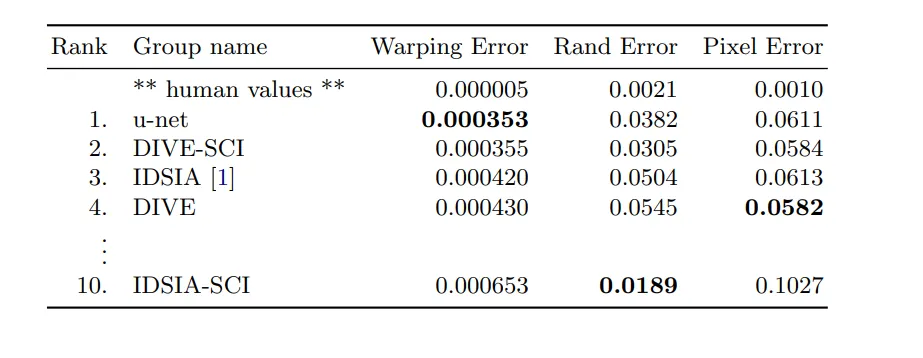
4. Experiments
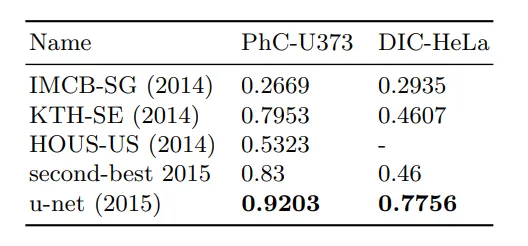
Em segmentation challenge
Isbi cell tracking challenge
5. Conclusion
biomedical image가 가지는 한계를 극복하기 위해 다양한 기법 도입 (매우 적은양의 데이터를 가지고도 학습가능)
•
데이터 셋의 부족
◦
overlap -tite strategy
◦
mirrrioring extrapolation
◦
elastic deformations
•
겹치는 세포 분리
◦
Touching Cells separtion
receptive field는 대상을 다양한 관점(다양한 스케일)에서 보겠다는 것.
ex) 나를 그냥 보는 것과 쌍안경을 끼고 보는 것과 천체 망원경으로 보는 것…
왜 깊게 쌓느냐에 대한 의문도 풀림.
깊게 쌓으면서 다양하게 많이 보면 대상에 대한 특징을 더 잘 추출할 수 있음.