목차
1. 데이터 설명 및 보기
1-1. 데이터 불러오기
•
변수 정보
1.
Pregnancies : 임신 횟수
2.
Glucose : 혈당 (2시간 짜리 경구 포도당 내성 테스트 기준)
3.
BloodPressure : 혈압
4.
SkinThickness : 피부의 두께
5.
Insulin : 인슐린 주사를 맞은 횟수로 해석함
6.
BMI : 체질량지수
7.
DiabetesPedigreeFunction : 당뇨병 혈통 기능...? 당뇨병의 유전성과 관련이 있는 것 같음
8.
Age : 나이
9.
Outcome : 결과 (768개 데이터 중 268개는 1 나머지는 0)
import pandas as pd
import matplotlib as mpl
import matplotlib.pylab as plt
import seaborn as sns
import numpy as np
file_path = 'Diabetes/diabetes.csv'
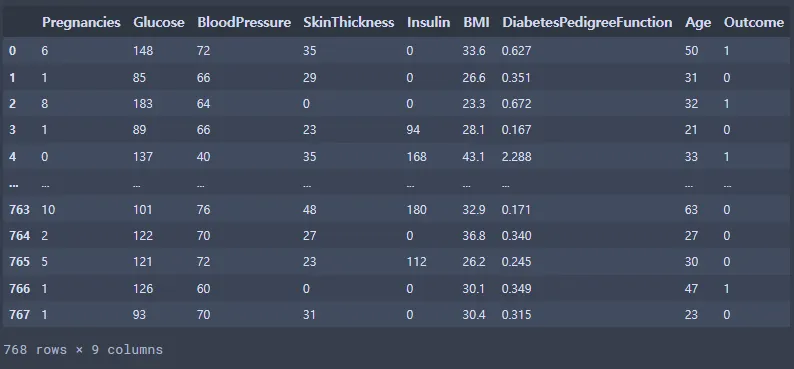
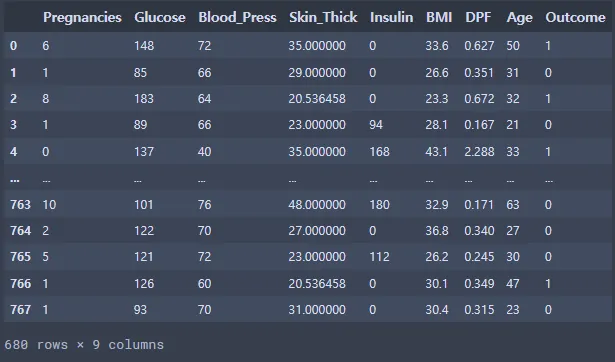
di_data = pd.read_csv(file_path)
di_data
Python
복사
1-2. 데이터 백업
di_data_copy_backup = di_data.copy()
di_data.to_csv('Diabetes/diabetes_backup.csv')
file_path = 'Diabetes/diabetes_backup.csv'
di_data_csv_backup = pd.read_csv(file_path)
di_data_csv_backup
Python
복사
di_data.info()
Python
복사
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 768 entries, 0 to 767
Data columns (total 9 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Pregnancies 768 non-null int64
1 Glucose 768 non-null int64
2 BloodPressure 768 non-null int64
3 SkinThickness 768 non-null int64
4 Insulin 768 non-null int64
5 BMI 768 non-null float64
6 DiabetesPedigreeFunction 768 non-null float64
7 Age 768 non-null int64
8 Outcome 768 non-null int64
dtypes: float64(2), int64(7)
memory usage: 54.1 KB
Plain Text
복사
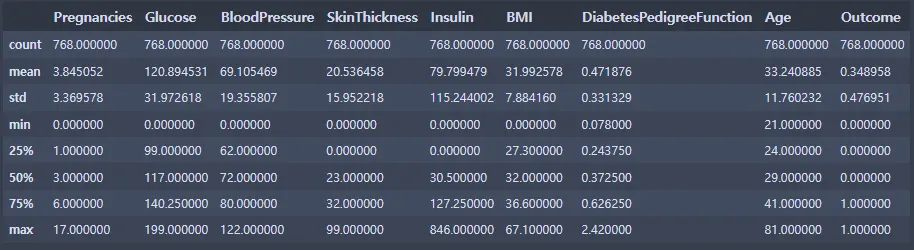
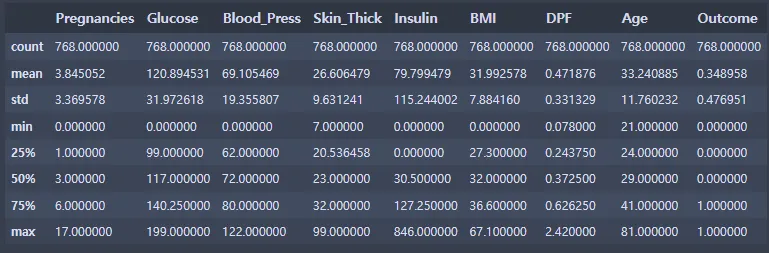
di_data.describe()
Python
복사
2. 데이터 기초 분석 및 탐색
2-1. 데이터 재구조화
di_data2 = di_data.rename({'BloodPressure':'Blood_Press', 'SkinThickness':'Skin_Thick', 'DiabetesPedigreeFunction':'DPF'}, axis = 'columns') #열의 이름 수정
di_data2
Python
복사
변수 이름 간략화
3. 데이터 클린징
3-1. 결측치 확인
import missingno as msno
di_data2.isnull().sum()
Python
복사
Pregnancies 0
Glucose 0
Blood_Press 0
Skin_Thick 0
Insulin 0
BMI 0
DPF 0
Age 0
Outcome 0
dtype: int64
Plain Text
복사
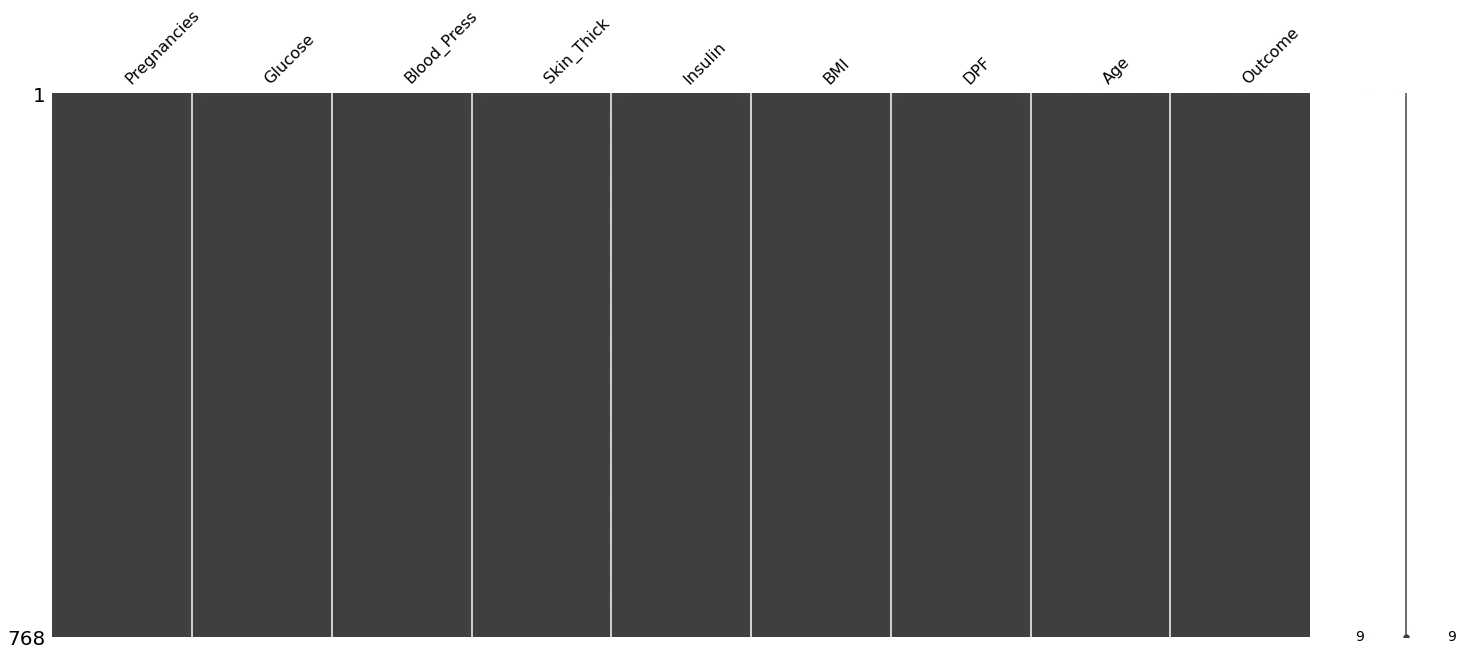
msno.matrix(di_data2)
Python
복사
Skin_Thick와 Insulin의 0값을 삭제하고 싶었는데 Insulin은 데이터간의 편차가 꽤 크고 0인 값이 너무 많아서 그냥 둠.
Skin_Thick는 평균값으로 대체했다.
di_data2.loc[di_data2['Skin_Thick']==0, 'Skin_Thick']=di_data2['Skin_Thick'].mean()
Python
복사
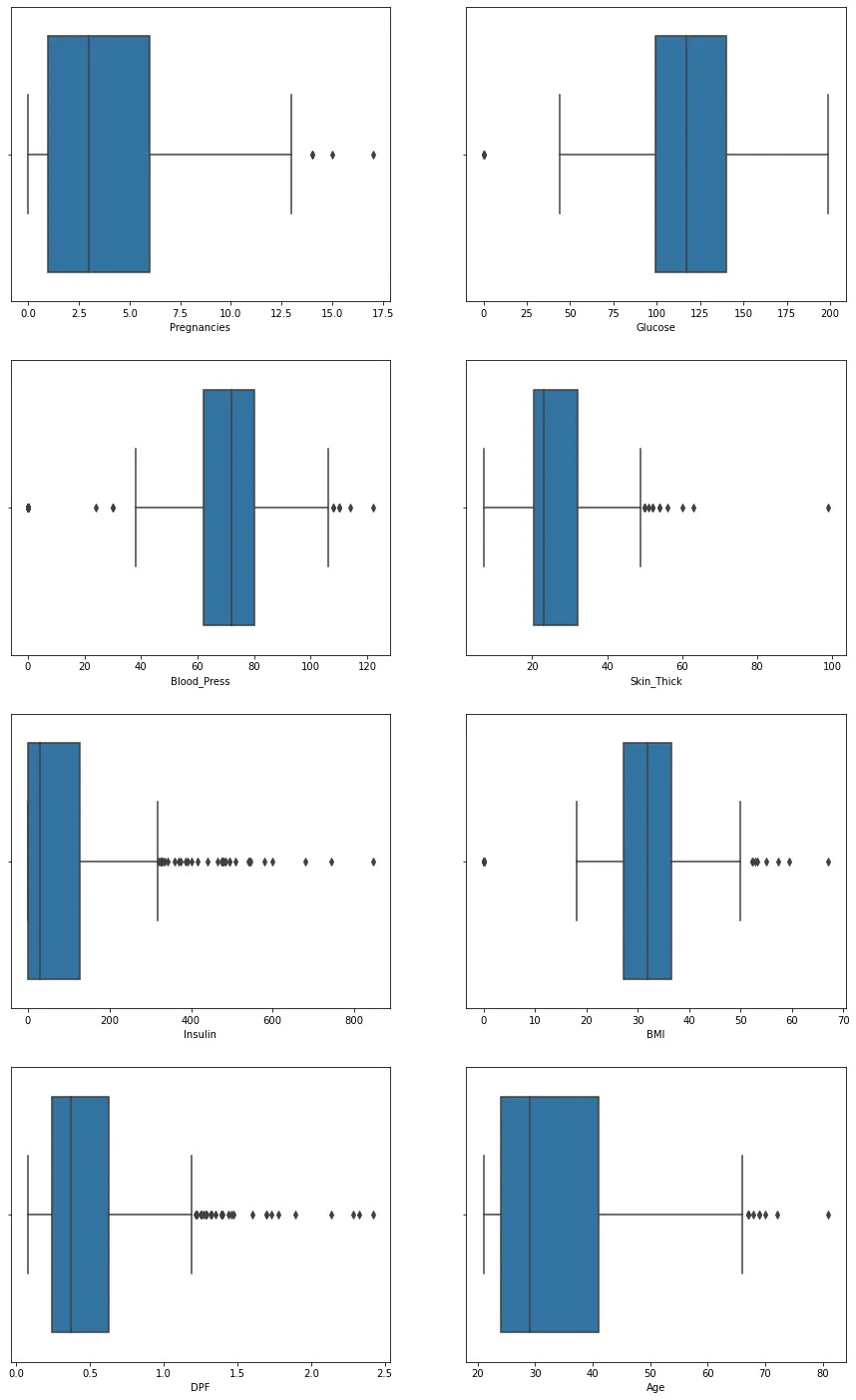
3-2. 이상치 확인
figure, ((ax1, ax2), (ax3, ax4), (ax5, ax6), (ax7, ax8)) = plt.subplots(nrows=4, ncols=2) #표를 넣을 4칸 생성
figure.set_size_inches(15, 25) # 각 표의 사이즈 정하기
sns.boxplot(x = "Pregnancies", data = di_data2, ax=ax1)
sns.boxplot(x = "Glucose", data = di_data2, ax=ax2)
sns.boxplot(x = "Blood_Press", data = di_data2, ax=ax3)
sns.boxplot(x = "Skin_Thick", data = di_data2, ax=ax4)
sns.boxplot(x = "Insulin", data = di_data2, ax=ax5)
sns.boxplot(x = "BMI", data = di_data2, ax=ax6)
sns.boxplot(x = "DPF", data = di_data2, ax=ax7)
sns.boxplot(x = "Age", data = di_data2, ax=ax8)
Python
복사
Insulin과 DPF는 유의미한 결과가 있을것으로 판단하여 이상치 분석. 나머지는 이상치 모두 제거.
Insulin은 600이상 제거, DPF는 이상치를 제거하지 않고 분석.
di_data2.describe()
Python
복사
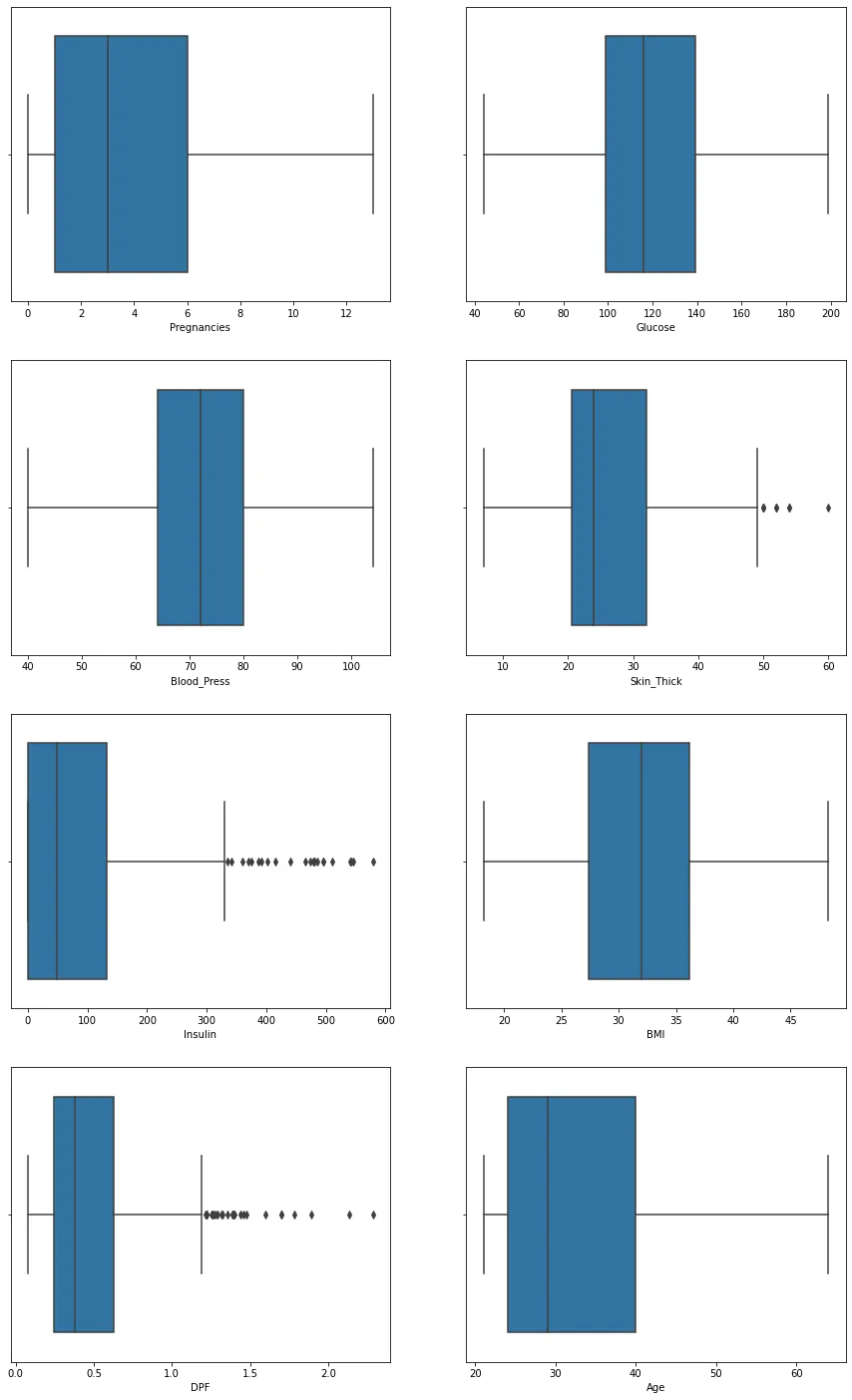
di_data3 = di_data2[(di_data2["Insulin"]<600) & (di_data2["Pregnancies"]<14) & (di_data2["Glucose"]>0) & (di_data2["Blood_Press"]<106) & (di_data2["Blood_Press"]>38) & (di_data2["Skin_Thick"]<90) & (di_data2["BMI"]<49) & (di_data2["BMI"]>0) & (di_data2["Age"]<65)]
di_data3
Python
복사
figure, ((ax1, ax2), (ax3, ax4), (ax5, ax6), (ax7, ax8)) = plt.subplots(nrows=4, ncols=2) #표를 넣을 4칸 생성
figure.set_size_inches(15, 25) # 각 표의 사이즈 정하기
sns.boxplot(x = "Pregnancies", data = di_data3, ax=ax1)
sns.boxplot(x = "Glucose", data = di_data3, ax=ax2)
sns.boxplot(x = "Blood_Press", data = di_data3, ax=ax3)
sns.boxplot(x = "Skin_Thick", data = di_data3, ax=ax4)
sns.boxplot(x = "Insulin", data = di_data3, ax=ax5)
sns.boxplot(x = "BMI", data = di_data3, ax=ax6)
sns.boxplot(x = "DPF", data = di_data3, ax=ax7)
sns.boxplot(x = "Age", data = di_data3, ax=ax8)
Python
복사
Insulin 600 이상 / Pregnacies 14 이상 / Blood_Press 106 이상, 38 이하 / Skin_Thick 90 이상 / BMI 49 이상 / Age 65 이상 이상치를 모두 제거함.
4. 데이터 시각화
4-1. catplot
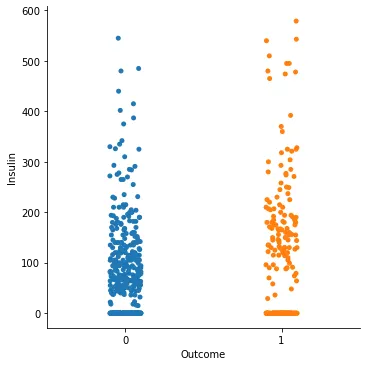
sns.catplot(x = "Outcome", y = "Insulin", data=di_data3)
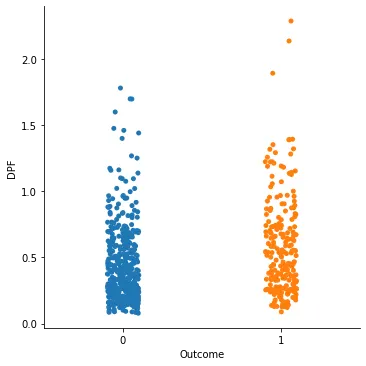
sns.catplot(x = "Outcome", y = "DPF", data=di_data3)
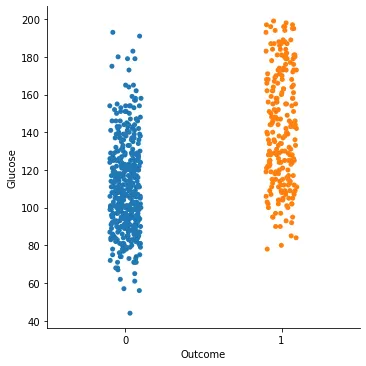
sns.catplot(x = "Outcome", y = "Glucose", data=di_data3)
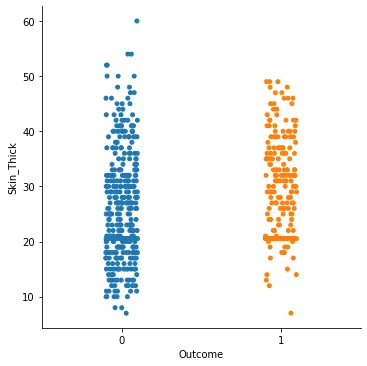
sns.catplot(x = "Outcome", y = "Skin_Thick", data=di_data3)
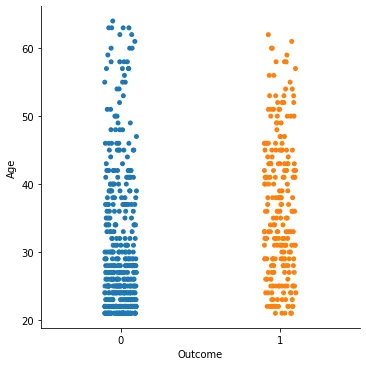
sns.catplot(x = "Outcome", y = "Age", data=di_data3)
Python
복사
Glucose는 뚜렷한 지표가 될 것 같고, Age와 Skin_Thick가 조금 차이를 보이는 것 이외에 다른 데이터들은 별로 의미가 없어보인다.
4-2. histogram
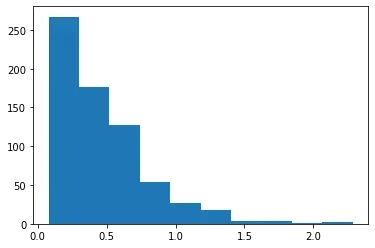
plt.hist(di_data3['DPF'])
plt.show()
Python
복사
DPF는 1이상인 값이 거의 없다. 데이터가 정확히 무엇을 의미하는지 모르겠다.
5. 상관분석
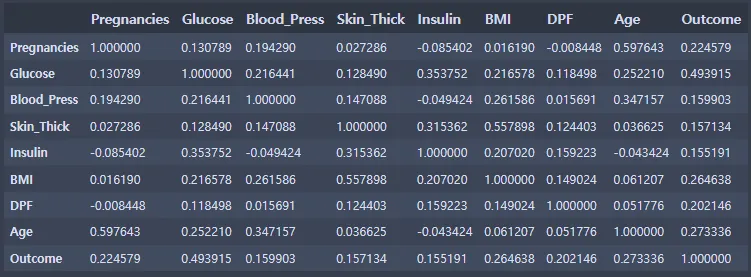
corrMatt = di_data3[['Pregnancies', 'Glucose', 'Blood_Press', 'Skin_Thick', 'Insulin', 'BMI', 'DPF', 'Age', 'Outcome']]
corrMatt = corrMatt.corr()
corrMatt
Python
복사
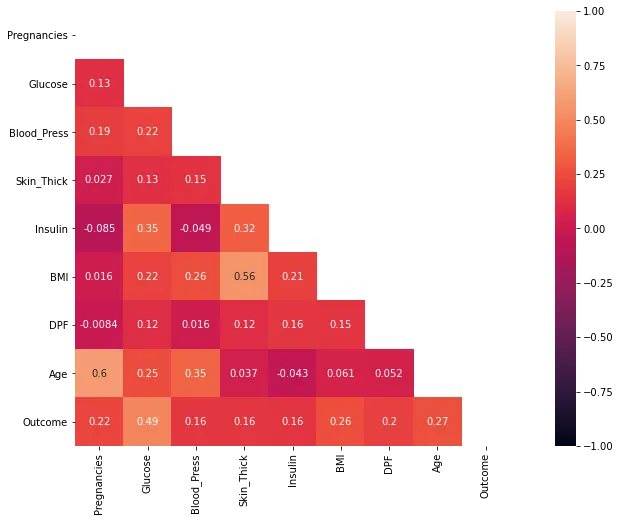
mask=np.zeros_like(corrMatt, dtype=np.bool)
mask[np.triu_indices_from(mask)] = True
figure, ax = plt.subplots()
figure.set_size_inches(16, 8)
sns.heatmap(corrMatt, mask=mask, vmin=-1, vmax=1, square=True, annot=True)
Python
복사
뇌졸중은 혈당과 가장 큰 상관관계를 이루고 나이, BMI, 임신 횟수, DPF와 약한 상관관계를 이룬다.
강한 상관관계인 데이터
•
나이와 임신횟수
뚜렷한 상관관계인 데이터
•
인슐린과 혈당
•
인슐린과 피부두께
•
BMI와 혈압
•
BMI와 피부두께
•
나이와 혈당
•
나이와 혈압
6. 회귀분석
•
종속 변수 : Outcome, Age
•
독립 변수 : Blood_press, Glucose
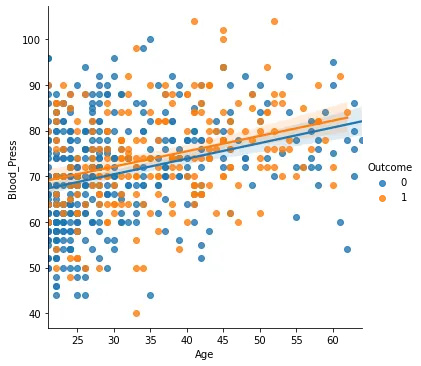
sns.lmplot(x = 'Age', y = 'Blood_Press', hue='Outcome', data=di_data3)
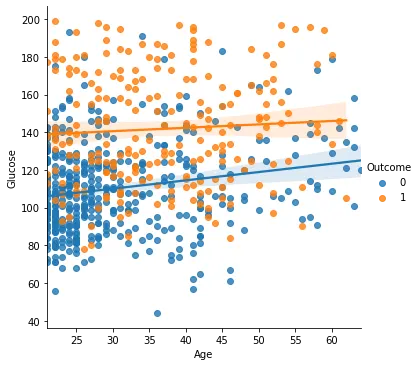
sns.lmplot(x = 'Age', y = 'Glucose', hue='Outcome', data=di_data3)
Python
복사
모두 양의 상관관계를 가지고 있고. Glucose에 따른 결과값이 꽤 차이가 난다.
혈당이 높을수록 당뇨병에 잘 걸린다.
•
종속 변수 : Outcome, BMI
•
독립 변수 : Blood_press, Skin_Thick
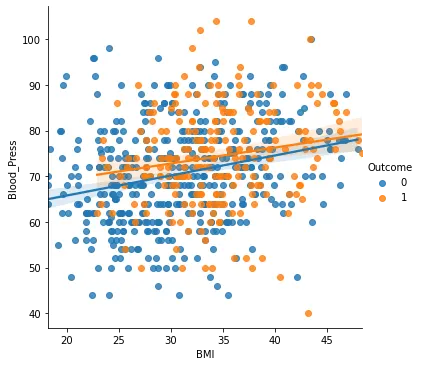
sns.lmplot(x = 'BMI', y = 'Blood_Press', hue='Outcome', data=di_data3)
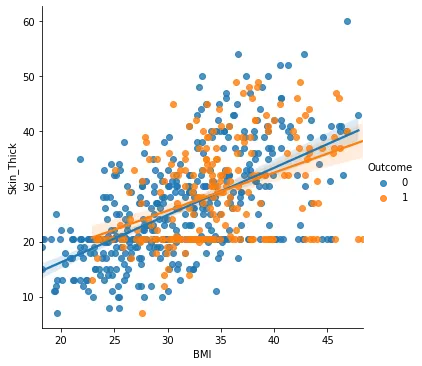
sns.lmplot(x = 'BMI', y = 'Skin_Thick', hue='Outcome', data=di_data3)
Python
복사
모두 양의 상관관계를 가지고 있지만 혈압과 피부두께 모두 당뇨와는 크게 상관이 없어 보인다.
•
종속 변수 : Outcome, Insulin
•
독립 변수 : Glucose, Skin_Thick
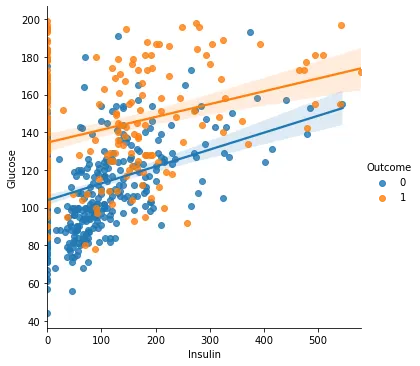
sns.lmplot(x = 'Insulin', y = 'Glucose', hue='Outcome', data=di_data3)
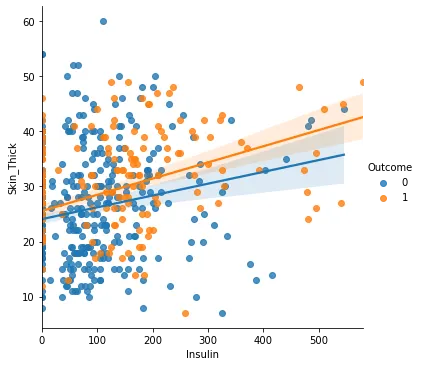
sns.lmplot(x = 'Insulin', y = 'Skin_Thick', hue='Outcome', data=di_data3)
Python
복사
모두 양의 상관관계를 가지고 있고 인슐린에 따른 피부두께는 뚜렷한 관계를 보인다. 혈당은 위의 결과와 같이 당뇨와 관련이 있어보인다.
결론 : 혈당(Glucose)은 당뇨와 매우 큰 관련이 있다. 혈당이 높은 사람은 당뇨에 걸릴 확률이 높다. 인슐린과 피부 두께도 어느정도 영향을 미친다.
추가적으로 얻은 정보 : 인슐린(Insulin) 수치가 증가함에 따라 피부의 두께도 두꺼워진다, BMI가 높아질수록 혈압, 피부의 두께가 두꺼워진다, 나이가 많아질수록 혈압이 높아진다.